手把手教你组织数据科学项目!(附代码)
为什么要重视项目结构?
说起数据分析,我们往往会想到报告结果、深入见解或可视化。通常这些最终结果占据了主要地位,所以人们很容易专注于让结果看起来漂亮而忽略了生成它们的代码质量。但是,这些最终结果都是以编程方式创建的,所以代码质量仍然很重要!此处我们讨论的不是代码缩进或格式标准,数据科学的代码质量的最终标准是正确性和可再现性(reproducibility)。
众所周知,好的分析通常是随意和偶然探索的结果。各种没得到结果的探索性实验和快速测试都是通往好结果道路上的一部分,并且没有灵丹妙药可以将数据探索转变为简单的线性过程。
一旦开始一个项目,就很难再去思考代码结构和项目布局了。所以最好从一个干净、合乎逻辑的结构开始并一以贯之。我们认为使用这样的标准化设置是非常有好处的。原因如下:
其他人会感谢你
定义明确的标准项目结构意味着新手无需研究大量文档就可以理解一项分析。这也意味着他们不一定非得阅读100%的代码才知道去哪里找特定的内容。
组织良好的代码往往能做到自我记录(self-documenting),组织结构本身可以在无需太多开销的情况下为你的代码提供上下文。人们会因此感谢你,因为他们可以:
● 更轻松地与你在此分析中合作
● 从你对流程和领域的分析中学习
● 对分析得到的结论充满信心
你可以在任何主流的Web开发框架(如Django或Ruby on Rails)中找到这方面的好例子。在创建一个新的Rails项目之前,没有人会去思考要在哪个位置放什么,他们只是运行rails new来获得像其他人一样的标准项目骨架(skeleton)。由于该默认结构在大多数项目中都是合乎逻辑且合理的,因此从未见过该特定项目的人也可以容易地找到各种部件。
另一个很好的例子是类Unix系统的文件系统层次结构标准(Filesystem Hierarchy Standard)。/ etc目录有非常特定的目的,就像/ tmp文件夹一样,每个人(或多或少)都同意遵守该约定。这意味着Red Hat用户和Ubuntu用户都知道在哪里查找某些类型的文件,即使他们使用的是对方的系统或者其他任何符合该标准的系统!
理想情况下,当同事打开您的数据科学项目时,也应该如此。
你会感谢自己
你尝试过重现几个月前甚至几年前做的分析吗?你可能编写了代码,但现在却不知道是否应该使用make_figures.py.old,make_figures_working.py或者new_make_figures01.py来完成工作。以下是我们的一些问题:
● 在开始之前,是否应该主动把X列加入到数据中?还是说其中某个notebook可以完成这一步?
● 想一想,在运行绘图代码之前我们必须首先运行哪个notebook:它是“过程数据”还是“干净数据”?
● 地理图形中所用的shapefiles是从哪下载的?
● 等类似问题
这些问题让人头疼,并且是无组织项目的症状。良好的项目结构应该让人很容易回到旧时的工作,例如分离关注点,将分析抽象为DAG,以及版本控制等实践。
没有任何约束
不赞同某些默认文件夹名称?正在做一个不标准且与当前结构不完全匹配的项目?更愿意使用与(少数)默认包不同的包?
去吧!这是一种轻量级结构,旨在成为许多项目的良好起点。正如PEP 8所说:
项目内部的一致性更为重要。一个模块或功能内部的一致性是最重要的。...但是,要知道何时不一致,有时风格指南建议并不适用。如有疑问,请用你的最佳判断。查看其他示例并确定最佳效果。并且不要犹豫去提问!
开始
我们为Python项目创建了一个数据科学cookiecutter模板。您的分析不一定要在Python中,但模板确实提供了一些Python样板,您可能想要删除它们(例如,在src文件夹中,以及docs中的Sphinx文档框架)。
需求
● Python 2.7 或 3.5
● cookiecutter Python package >= 1.4.0: pip install cookiecutter
开始一个新项目
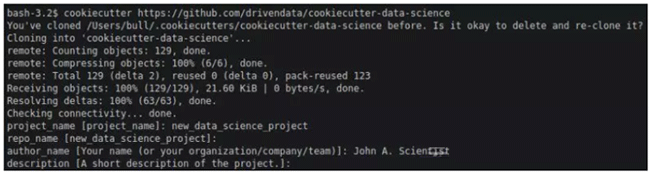
启动新项目只需在命令行中运行如下命令。无需先创建目录,cookiecutter将为您完成。
cookiecutter
https://github.com/drivendata/cookiecutter-data-science
示例
原文中是一段视频,建议从原文链接中观看。
原文链接:
https://www.kdnuggets.com/2018/07/cookiecutter-data-science-organize-data-project.html
目录结构
├── LICENSE
├── Makefile <- Makefile with commands like `make data` or `make train`
├── README.md <- The top-level README for developers using this project.
├── data
│ ├── external <- Data from third party sources.
│ ├── interim <- Intermediate data that has been transformed.
│ ├── processed <- The final, canonical data sets for modeling.
│ └── raw <- The original, immutable data dump.
│
├── docs <- A default Sphinx project; see sphinx-doc.org for details
│
├── models <- Trained and serialized models, model predictions, or model summaries
│
├── notebooks <- Jupyter notebooks. Naming convention is a number (for ordering),
│ the creator's initials, and a short `-` delimited description, e.g.
│ `1.0-jqp-initial-data-exploration`.
│
├── references <- Data dictionaries, manuals, and all other explanatory materials.
│
├── reports <- Generated analysis as HTML, PDF, LaTeX, etc.
│ └── figures <- Generated graphics and figures to be used in reporting
│
├── requirements.txt <- The requirements file for reproducing the analysis environment, e.g.
│ generated with `pip freeze > requirements.txt`
│
├── src <- Source code for use in this project.
│ ├── __init__.py <- Makes src a Python module
│ │
│ ├── data <- Scripts to download or generate data
│ │ └── make_dataset.py
│ │
│ ├── features <- Scripts to turn raw data into features for modeling
│ │ └── build_features.py
│ │
│ ├── models <- Scripts to train models and then use trained models to make
│ │ │ predictions
│ │ ├── predict_model.py
│ │ └── train_model.py
│ │
│ └── visualization <- Scripts to create exploratory and results oriented visualizations
│ └── visualize.py
│
└── tox.ini <- tox file with settings for running tox; see tox.testrun.org
观点
项目结构中隐含着一些观点,这些观点源于我们在数据科学项目合作时可行的和不可行的工作经验。一些观点是关于工作流程的,一些是关于提高效率的工具的。以下是该项目所依据的一些信念,如果您有想法,请提供或分享。
数据是不可变的
不要编辑原始数据,尤其不要手动编辑,也不要在Excel中编辑。不要覆盖原始数据。不要保存多个版本的原始数据。将数据(及其格式)视为不可变的。你编写的代码应该让原始数据通过分析流程管道并得到最终分析。没必要每次想要创建一个新计算时都运行所有步骤(请参阅Analysis是DAG),但是任何人都应该能够仅使用src中的代码和data /raw中的数据来重现分析结果。
此外,如果数据是不可变的,则它不需要像代码那样进行源代码控制。因此,默认情况下,data文件夹包含在.gitignore文件中。如果你有少量很少会被更改的数据,你可能希望将数据包含在repo中。目前Github会警告文件是否超过50MB并且拒绝超过100MB的文件。用于存储/同步大型数据的一些其他方案包括同步工具(例如,s3cmd的AWS S3,Git Large File Storage,Git Annex和dat)。目前默认情况下,我们请求一个S3 bucket并使用AWS CLI将data文件夹中的数据与服务器同步。
Notebooks用于探索和交流
Jupyter notebook, Beaker notebook, Zeppelin和其他交互式编程工具对于探索性数据分析(EDA)来说是非常高效的。然而,这些工具对重现分析而言效果较差。 当我们在工作中使用notebook时,我们经常细分notebook的文件夹。例如,notebook/exploration包括初始探索,而notebook/report包含更漂亮的工作,并可以作为html格式导出到report目录。
由于notebooks挑战了源代码控制的目标(例如,json的差异通常不是人类可读的,并且几乎不可能合并),我们不建议直接在Jupyter notebooks上与其他人合作。为了有效的使用notebooks,这里有两个建议:
● 遵守一种命名习惯,可以展现作者以及所做分析的顺序。我们使用这种格式:
● 重构某些好的部分。完成相同任务的代码不要写在多个notebook中。如果是一个数据预处理任务,放入处理管道并把代码写在src/data/make_dataset.py中,并且从data/interim中加载数据。如果是有用的工具代码,重构到src中。
译者注:用文本编辑器打开jupyter notebook的文件,可以看到其本质上是一个json格式的文件,所以这里作者说很难使用Git去diff或merge不同版本的notebook代码,不过目前已有工具致力于解决这个问题,例如nbdime。
默认情况我们把项目转换成一个python包(见setup.py文件)。你可以导入自己的代码,并且在notebook中使用,方法如下:
# OPTIONAL: Load the "autoreload" extension so that code can change
%load_ext autoreload
# OPTIONAL: always reload modules so that as you change code in src, it gets loaded
%autoreload 2
from src.data import make_dataset
分析就是DAG
在分析中,你常常会有一些运行时间很长的步骤,比如预处理数据或者训练模型。如果这些步骤已经运行过了(并且你已经将输出保存在了某处,例如data/interim目录),那你肯定不想每次都等待它们重新运行。我们更喜欢用make来管理彼此依赖的步骤,尤其是运行时间很长的步骤。Make是基于Unix的平台上的常用工具(可用于Windows)。遵守make官方文档, Makefile conventions和portability guide将帮助确保你的Makefile在各个系统中有效工作。这是一些入门示例。许多从事数据工作的人都使用make作为他们的首选工具,其中就包括Mike Bostock。
还有其他用来管理使用Python而不是DSL(领域特定语言)编写的DAGs的工具(例如,Paver,Luigi,Airflow,Snakemake,Ruffus或Joblib)。如果它们更适合您的分析,请随意使用。
从环境开始构建
重现某个分析的第一步通常是重现它运行的计算环境。你需要相同的工具,相同的库和相同的版本,以使一切都很好地协同工作。
一个有效的方法是使用virtualenv(我们建议使用virtualenvwrapper来管理virtualenvs)。通过列举repo中的所有需求(我们包含一个requirements.txt文件),你可以轻松跟踪用于重现分析所需的包。以下是一个不错的工作流程:
● 运行mkvirtualenv创建新项目
● pip install分析所需的包
● 运行pip freeze > requirements.txt以确定用于重现分析的确切包版本
● 如果你发现需要安装其他软件包,请再次运行pip freeze > requirements.txt并将更改提交到版本控制。
如果你对重新创建环境有更复杂的要求,请考虑基于虚拟机的方法,如Docker或Vagrant。这两个工具都使用基于文本的格式(分别为Dockerfile和Vagrantfile),你可以轻松添加到源代码控制中,以描述如何创建满足需求的虚拟机。
密码和配置不要加入版本控制
你肯定不想在Github上泄露你的AWS密钥或Postgres用户名和密码。不再赘述,请看关于这一点的Twelve Factor App。这是一种方法:
将您的机密和配置变量存储在一个特殊文件中
在项目根目录中创建一个.env文件。感谢.gitignore,永远不要把该文件提交到版本控制repo中。以下是一个例子:
# example .env file
DATABASE_URL=postgres://username:password@localhost:5432/dbname
AWS_ACCESS_KEY=myaccesskey
AWS_SECRET_ACCESS_KEY=mysecretkey
OTHER_VARIABLE=something
使用包自动加载这些变量
如果查看src / data / make_dataset.py中的存根脚本,它使用了一个名为python-dotenv的包将此文件中的所有条目作为环境变量加载,以便可以使用os.environ.get访问它们。这是一个改编自python-dotenv文档的示例片段:
# src/data/dotenv_example.py
import os
from dotenv import load_dotenv, find_dotenv
# find .env automagically by walking up directories until it's found
dotenv_path = find_dotenv()
# load up the entries as environment variables
load_dotenv(dotenv_path)
database_url = os.environ.get("DATABASE_URL")
other_variable = os.environ.get("OTHER_VARIABLE")
AWS CLI配置
当使用Amazon S3存储数据时,有一个简单的方法来管理AWS访问,即把访问keys设置为环境变量。但是在一台机器上管理多个keys(例如,同时有多个项目)时,最好还是使用一个凭证文件(credentials file),通常放在~/.aws/credentials,该文件看起来像这样:
[default]
aws_access_key_id=myaccesskey
aws_secret_access_key=mysecretkey
[another_project]
aws_access_key_id=myprojectaccesskey
aws_secret_access_key=myprojectsecretkey
你可以在初始化项目时添加名称。假设未设置合适的环境变量,则使用默认的配置。
保守地更改默认文件夹结构
为了使这种结构广泛适用于不同类型的项目,我们认为最好的方法是对于你自己的项目而言,你可以自由更改文件夹,但在改变用于所有项目的默认结构时要保守。
我们专门为添加,删减,重命名或移动文件夹的问题创建了一个文件夹布局标签。 更一般地说,我们还创建了一个需求讨论标签,用于在实施之前应该进行仔细讨论和广泛支持的问题。
贡献
Cookiecutter数据科学项目有点武断,但不怕出错。最佳实践总在改变,工具一直在发展,我们从中吸取了教训。该项目的目标是使启动、构建和共享一个分析更加容易。我们鼓励提出请求和提交问题。我们很想知道什么对你有用,什么没用。
如果你使用Cookiecutter数据科学项目,请链接回此页面或给我们一个赞,并让我们知道!
相关项目和参考链接
项目结构和可再现性更多地在R研究社区中被讨论。以下是一些项目和博客文章,如果你使用R工作则可能会帮到你。
● Project Template - An R data analysis template
● "Designing projects" on Nice R Code
● "My research workflow" on Carlboettifer.info
● "A Quick Guide to Organizing Computational Biology Projects" in PLOS Computational Biology
最后,非常感谢Cookiecutter项目(github),它帮助我们花更少的时间思考和编写样板文件,从而花更多的时间去完成工作。

时间:2018-10-12 22:24 来源: 转发量:次
声明:本站部分作品是由网友自主投稿和发布、编辑整理上传,对此类作品本站仅提供交流平台,转载的目的在于传递更多信息及用于网络分享,并不代表本站赞同其观点和对其真实性负责,不为其版权负责。如果您发现网站上有侵犯您的知识产权的作品,请与我们取得联系,我们会及时修改或删除。
相关文章:
相关推荐:
网友评论: